| Autores: | Emmanuelle Gouillart, Gaël Varoquaux |
|---|
Imagen = array numérico en 2-D (o 3-D: TC, IRM, 2D + tiempo; 4-D, ...)
Aquí, imagen == array Numpy np.array
Herramientas usadas en este tutorial:
numpy: manipulación básica de arrays
scipy: scipy.ndimage submódulo dedicado a procesamiento de imágenes (imágenes n-dimensionales). Ver http://docs.scipy.org/doc/scipy/reference/tutorial/ndimage.html
>>> from scipy import ndimage
algunos ejemplos usan librerías especializadas que hacen uso de np.array:
Tareas comunes en procesamiento de imágenes:
Existen módulos más potentes y completos:
Contenido
Escribiendo un array a un archivo:
from scipy import misc
l = misc.lena()
misc.imsave('lena.png', l) # uses the Image module (PIL)
import matplotlib.pyplot as plt
plt.imshow(l)
plt.show()

Creando un array numpy desde un archivo de imagen
>>> lena = misc.imread('lena.png')
>>> type(lena)
<type 'numpy.ndarray'>
>>> lena.shape, lena.dtype
((512, 512), dtype('uint8'))
dtype es uint8 para imágenes de 8-bits (0-255)
Abriendo archivos raw (de cámara, imágenes 3-D)
>>> l.tofile('lena.raw') # Creación de un fichero raw
>>> lena_from_raw = np.fromfile('lena.raw', dtype=np.int64)
>>> lena_from_raw.shape
(262144,)
>>> lena_from_raw.shape = (512, 512)
>>> import os
>>> os.remove('lena.raw')
Necesitas saber la forma y el dtype (tipo de dato) de la imagen (de qué forma separar los bytes de datos).
Para conjuntos de datos de gran tamaño se puede usar np.memmap, que sirve para mapear en memoria estos datos
>>> lena_memmap = np.memmap('lena.raw', dtype=np.int64, shape=(512, 512))
(los datos son leídos desde el archivo, pero no son cargados en la memoria)
Trabajando en una lista de archivos de imágenes
>>> for i in range(10):
... im = np.random.random_integers(0, 255, 10000).reshape((100, 100))
... misc.imsave('random_%02d.png' % i, im)
>>> from glob import glob
>>> filelist = glob('random*.png')
>>> filelist.sort()
Se puede usar matplotlib e imshow para mostrar una imagen dentro de una figura matplotlib
>>> l = scipy.lena()
>>> import matplotlib.pyplot as plt
>>> plt.imshow(l, cmap=plt.cm.gray)
<matplotlib.image.AxesImage object at 0x3c7f710>
Podemos incrementar el contraste ajustando los valores mínimos y máximos
>>> plt.imshow(l, cmap=plt.cm.gray, vmin=30, vmax=200)
<matplotlib.image.AxesImage object at 0x33ef750>
>>> # Remove axes and ticks
>>> plt.axis('off')
(-0.5, 511.5, 511.5, -0.5)
Podemos dibujar las líneas de contorno
>>> plt.contour(l, [60, 211])
<matplotlib.contour.ContourSet instance at 0x33f8c20>
Para hacer una inspección detallada de variaciones de intensidad podemos usar interpolation='nearest'
>>> plt.imshow(l[200:220, 200:220], cmap=plt.cm.gray)
>>> plt.imshow(l[200:220, 200:220], cmap=plt.cm.gray, interpolation='nearest')
A veces, otros paquetes usan paquetes o toolkits para visualización (GTK, Qt)
>>> import scikits.image.io as im_io
>>> im_io.use_plugin('gtk', 'imshow')
>>> im_io.imshow(l)
Visualización en 3-D: Mayavi
Ver Gráficos 3D con Mayavi y Datos volumétricos.

Las imágenes las leemos usando arrays numpy: ¡Podemos usar toda la maquinaria numpy!

>>> lena = scipy.lena()
>>> lena[0, 40]
166
>>> # Slicing
>>> lena[10:13, 20:23]
array([[158, 156, 157],
[157, 155, 155],
[157, 157, 158]])
>>> lena[100:120] = 255
>>>
>>> lx, ly = lena.shape
>>> X, Y = np.ogrid[0:lx, 0:ly]
>>> mask = (X - lx/2)**2 + (Y - ly/2)**2 > lx*ly/4
>>> # Masks
>>> lena[mask] = 0
>>> # Fancy indexing
>>> lena[range(400), range(400)] = 255
>>> lena = scipy.lena()
>>> lena.mean()
124.04678344726562
>>> lena.max(), lena.min()
(245, 25)
np.histogram
Ejercicio 1

>>> lena = scipy.lena()
>>> lx, ly = lena.shape
>>> # Recorte de la imagen
>>> crop_lena = lena[lx/4:-lx/4, ly/4:-ly/4]
>>> # up <-> down flip
>>> flip_ud_lena = np.flipud(lena)
>>> # rotación
>>> rotate_lena = ndimage.rotate(lena, 45)
>>> rotate_lena_noreshape = ndimage.rotate(lena, 45, reshape=False)
Filtros locales: reemplaza el valor de los pixeles por una función de los valores de los pixeles vecinos.
Vecindad: cuadrado (selecciona el tamaño), círculo o elementos estructurados más complejos*.

Filtro gaussiano de scipy.ndimage
>>> from scipy import misc
>>> lena = misc.lena()
>>> blurred_lena = ndimage.gaussian_filter(lena, sigma=3)
>>> very_blurred = ndimage.gaussian_filter(lena, sigma=5)
Filtro uniforme
>>> local_mean = ndimage.uniform_filter(lena, size=11)
Enfocar una imagen borrosa
>>> from scipy import misc
>>> lena = misc.lena()
>>> blurred_l = ndimage.gaussian_filter(lena, 3)
Incrementamos el peso de los bordes agregando una aproximación del laplaciano
>>> filter_blurred_l = ndimage.gaussian_filter(blurred_l, 1)
>>> alpha = 30
>>> sharpened = blurred_l + alpha * (blurred_l - filter_blurred_l)
Imagen de Lena con ruido
>>> from scipy import misc
>>> l = misc.lena()
>>> l = l[230:310, 210:350]
>>> noisy = l + 0.4*l.std()*np.random.random(l.shape)
Un filtro gaussiano suaviza la imagen eliminando el ruido... además de los bordes
>>> gauss_denoised = ndimage.gaussian_filter(noisy, 2)
La mayoría de los filtros lineales isotrópicos locales (ndimage.uniform_filter) desenfocan la imagen.
Un filtro de mediana conserva mejor los bordes
>>> med_denoised = ndimage.median_filter(noisy, 3)
Filtro de mediana: mejor resultado para zonas de frontera rectas (baja curvatura)
>>> im = np.zeros((20, 20))
>>> im[5:-5, 5:-5] = 1
>>> im = ndimage.distance_transform_bf(im)
>>> im_noise = im + 0.2*np.random.randn(*im.shape)
>>> im_med = ndimage.median_filter(im_noise, 3)
Otros filtros de rango: ndimage.maximum_filter, ndimage.percentile_filter
Otros filtros no lineales locales: Wiener (scipy.signal.wiener), etc.
Filtros no locales
Reducción de ruido de variación total (TV, Total Variation). Encuentra una nueva imagen donde la variación total de la imagen (integral de la norma L1 del gradiente) es minimizada al avez que el resultado se mantiene cercano a la imagen medida
>>> from skimage.filter import tv_denoise
>>> tv_denoised = tv_denoise(noisy, weight=10)
>>> # More denoising (to the expense of fidelity to data)
>>> tv_denoised = tv_denoise(noisy, weight=50)
Ejercicio 2: Reducción de ruido
Ver http://en.wikipedia.org/wiki/Mathematical_morphology
Prueba una imagen con una forma simple (un elemento con estructura) y modifica esta imagen de acuerdo a como se ajuste o no a la imagen.
Elemento con estructura
>>> el = ndimage.generate_binary_structure(2, 1)
>>> el
array([[False, True, False],
[ True, True, True],
[False, True, False]], dtype=bool)
>>> el.astype(np.int)
array([[0, 1, 0],
[1, 1, 1],
[0, 1, 0]])
Erosión = filtro mínimo. Reemplaza el valor de un pixel por el valor mínimo del elemento estructurado
>>> a = np.zeros((7,7), dtype=np.int)
>>> a[1:6, 2:5] = 1
>>> a
array([[0, 0, 0, 0, 0, 0, 0],
[0, 0, 1, 1, 1, 0, 0],
[0, 0, 1, 1, 1, 0, 0],
[0, 0, 1, 1, 1, 0, 0],
[0, 0, 1, 1, 1, 0, 0],
[0, 0, 1, 1, 1, 0, 0],
[0, 0, 0, 0, 0, 0, 0]])
>>> ndimage.binary_erosion(a).astype(a.dtype)
array([[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 1, 0, 0, 0],
[0, 0, 0, 1, 0, 0, 0],
[0, 0, 0, 1, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0]])
>>> #Erosion elimina objectos más pequeños que la estructura
>>> ndimage.binary_erosion(a, structure=np.ones((5,5))).astype(a.dtype)
array([[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0]])
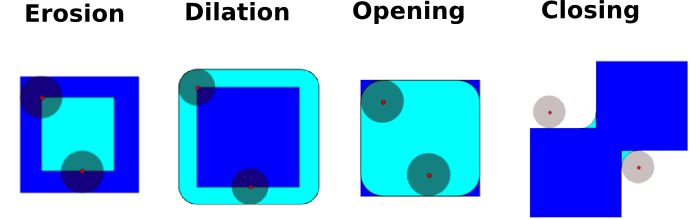
Dilatación: filtro máximo
>>> a = np.zeros((5, 5))
>>> a[2, 2] = 1
>>> a
array([[ 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0.],
[ 0., 0., 1., 0., 0.],
[ 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0.]])
>>> ndimage.binary_dilation(a).astype(a.dtype)
array([[ 0., 0., 0., 0., 0.],
[ 0., 0., 1., 0., 0.],
[ 0., 1., 1., 1., 0.],
[ 0., 0., 1., 0., 0.],
[ 0., 0., 0., 0., 0.]])
También funciona para imágenes en escala de grises
>>> np.random.seed(2)
>>> x, y = (63*np.random.random((2, 8))).astype(np.int)
>>> im[x, y] = np.arange(8)
>>> bigger_points = ndimage.grey_dilation(im, size=(5, 5), structure=np.ones((5, 5)))
>>> square = np.zeros((16, 16))
>>> square[4:-4, 4:-4] = 1
>>> dist = ndimage.distance_transform_bf(square)
>>> dilate_dist = ndimage.grey_dilation(dist, size=(3, 3), \
... structure=np.ones((3, 3)))
Opening (Apertura): erosión + dilatación
>>> a = np.zeros((5,5), dtype=np.int)
>>> a[1:4, 1:4] = 1; a[4, 4] = 1
>>> a
array([[0, 0, 0, 0, 0],
[0, 1, 1, 1, 0],
[0, 1, 1, 1, 0],
[0, 1, 1, 1, 0],
[0, 0, 0, 0, 1]])
>>> # Opening removes small objects
>>> ndimage.binary_opening(a, structure=np.ones((3,3))).astype(np.int)
array([[0, 0, 0, 0, 0],
[0, 1, 1, 1, 0],
[0, 1, 1, 1, 0],
[0, 1, 1, 1, 0],
[0, 0, 0, 0, 0]])
>>> # Opening can also smooth corners
>>> ndimage.binary_opening(a).astype(np.int)
array([[0, 0, 0, 0, 0],
[0, 0, 1, 0, 0],
[0, 1, 1, 1, 0],
[0, 0, 1, 0, 0],
[0, 0, 0, 0, 0]])
Aplicación (Application): eliminar ruido
>>> square = np.zeros((32, 32))
>>> square[10:-10, 10:-10] = 1
>>> np.random.seed(2)
>>> x, y = (32*np.random.random((2, 20))).astype(np.int)
>>> square[x, y] = 1
>>> open_square = ndimage.binary_opening(square)
>>> eroded_square = ndimage.binary_erosion(square)
>>> reconstruction = ndimage.binary_propagation(eroded_square, mask=square)
Cierre (Closing): dilatación + erosión
Esqueletización (Skeletonization): reduce objetos a finas líneas de un píxel de grosor manteniendo la misma topología
Existen muchas otras operaciones matemáticas de morfología: hit and miss transform, tophat, etc.
Datos sintéticos
>>> im = np.zeros((256, 256))
>>> im[64:-64, 64:-64] = 1
>>>
>>> im = ndimage.rotate(im, 15, mode='constant')
>>> im = ndimage.gaussian_filter(im, 8)
Usa un operador gradiente (Sobel) para encontrar altas variaciones de intensidad
>>> sx = ndimage.sobel(im, axis=0, mode='constant')
>>> sy = ndimage.sobel(im, axis=1, mode='constant')
>>> sob = np.hypot(sx, sy)
Filtro de Canny
>>> from skimage.filter import canny
>>> im += 0.1*np.random.random(im.shape)
>>> edges = canny(im, 1, 0.4, 0.2) # not enough smoothing
>>> edges = canny(im, 3, 0.3, 0.2) # better parameters
Se necesitan ajustar varios parámetros... riesgo de sobreajuste.
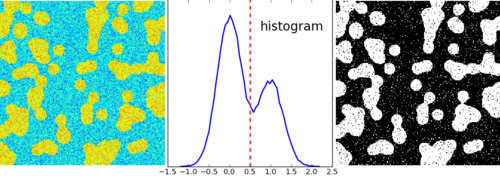
Segmentación basada en histograma (sin información espacial)
>>> n = 10
>>> l = 256
>>> im = np.zeros((l, l))
>>> np.random.seed(1)
>>> points = l*np.random.random((2, n**2))
>>> im[(points[0]).astype(np.int), (points[1]).astype(np.int)] = 1
>>> im = ndimage.gaussian_filter(im, sigma=l/(4.*n))
>>> mask = (im > im.mean()).astype(np.float)
>>> mask += 0.1 * im
>>> img = mask + 0.2*np.random.randn(*mask.shape)
>>> hist, bin_edges = np.histogram(img, bins=60)
>>> bin_centers = 0.5*(bin_edges[:-1] + bin_edges[1:])
>>> binary_img = img > 0.5
Umbral automático: usa el modelo de mezcla gaussiano
>>> mask = (im > im.mean()).astype(np.float)
>>> mask += 0.1 * im
>>> img = mask + 0.3*np.random.randn(*mask.shape)
>>> from sklearn.mixture import GMM
>>> classif = GMM(n_components=2)
>>> classif.fit(img.reshape((img.size, 1)))
GMM(...)
>>> classif.means
array([[ 0.9353155 ],
[-0.02966039]])
>>> np.sqrt(classif.covarsi).ravel()
array([ 0.35074631, 0.28225327])
>>> classif.weights
array([ 0.40989799, 0.59010201])
>>> threshold = np.mean(classif.means)
>>> binary_img = img > threshold
Usamos morfología matemática para limpiar el resultado
>>> # Elimina pequeñas regiones blancas
>>> open_img = ndimage.binary_opening(binary_img)
>>> # Elimina el pequeño agujero negro
>>> close_img = ndimage.binary_closing(open_img)
Ejercicio
Revisa que las operaciones de reconstrucción (erosión + propagación) produzcan un mejor resultado que apertura/cierre (opening/closing)
>>> eroded_img = ndimage.binary_erosion(binary_img)
>>> reconstruct_img = ndimage.binary_propagation(eroded_img, mask=binary_img)
>>> tmp = np.logical_not(reconstruct_img)
>>> eroded_tmp = ndimage.binary_erosion(tmp)
>>> reconstruct_final = np.logical_not(ndimage.binary_propagation(eroded_tmp, mask=tmp))
>>> np.abs(mask - close_img).mean()
0.014678955078125
>>> np.abs(mask - reconstruct_final).mean()
0.0042572021484375
Ejercicio
Revisa cómo un primer paso de reducción de ruido (filtro de mediana, variación total) modifica el histograma y revisa que la segmentación basada en histograma resultante es más exacta.
>>> from skimage.morphology import watershed, is_local_maximum
>>>
>>> # Generate an initial image with two overlapping circles
>>> x, y = np.indices((80, 80))
>>> x1, y1, x2, y2 = 28, 28, 44, 52
>>> r1, r2 = 16, 20
>>> mask_circle1 = (x - x1)**2 + (y - y1)**2 < r1**2
>>> mask_circle2 = (x - x2)**2 + (y - y2)**2 < r2**2
>>> image = np.logical_or(mask_circle1, mask_circle2)
>>> # Now we want to separate the two objects in image
>>> # Generate the markers as local maxima of the distance
>>> # to the background
>>> from scipy import ndimage
>>> distance = ndimage.distance_transform_edt(image)
>>> local_maxi = is_local_maximum(distance, image, np.ones((3, 3)))
>>> markers = ndimage.label(local_maxi)[0]
>>> labels = watershed(-distance, markers, mask=image)
Segmentation Spectral clustering (cortes normalizados)
>>> from sklearn.feature_extraction import image
>>> from sklearn.cluster import spectral_clustering
>>> l = 100
>>> x, y = np.indices((l, l))
>>> center1 = (28, 24)
>>> center2 = (40, 50)
>>> center3 = (67, 58)
>>> center4 = (24, 70)
>>> radius1, radius2, radius3, radius4 = 16, 14, 15, 14
>>> circle1 = (x - center1[0])**2 + (y - center1[1])**2 < radius1**2
>>> circle2 = (x - center2[0])**2 + (y - center2[1])**2 < radius2**2
>>> circle3 = (x - center3[0])**2 + (y - center3[1])**2 < radius3**2
>>> circle4 = (x - center4[0])**2 + (y - center4[1])**2 < radius4**2
>>> # 4 círculos
>>> img = circle1 + circle2 + circle3 + circle4
>>> mask = img.astype(bool)
>>> img = img.astype(float)
>>> img += 1 + 0.2*np.random.randn(*img.shape)
>>> # Convierte la imagen en un gráfico con el valor del gradiente
>>> # en los bordes.
>>> graph = image.img_to_graph(img, mask=mask)
>>> # Usa una función decreciente del gradiente: we take it weakly
>>> # dependant from the gradient the segmentation is close to a voronoi
>>> graph.data = np.exp(-graph.data/graph.data.std())
>>> labels = spectral_clustering(graph, k=4, mode='arpack')
>>> label_im = -np.ones(mask.shape)
>>> label_im[mask] = labels

Synthetic data
>>> n = 10
>>> l = 256
>>> im = np.zeros((l, l))
>>> points = l*np.random.random((2, n**2))
>>> im[(points[0]).astype(np.int), (points[1]).astype(np.int)] = 1
>>> im = ndimage.gaussian_filter(im, sigma=l/(4.*n))
>>> mask = im > im.mean()
Análisis de componentes conectados
Etiqueta de componentes conectados: ndimage.label
>>> label_im, nb_labels = ndimage.label(mask)
>>> nb_labels # how many regions?
23
>>> plt.imshow(label_im)
<matplotlib.image.AxesImage object at ...>
Calcula tamaño, valor medio, etcétera, de cada región
>>> sizes = ndimage.sum(mask, label_im, range(nb_labels + 1))
>>> mean_vals = ndimage.sum(im, label_im, range(1, nb_labels + 1))
Limpia componentes conectados pequeños
>>> mask_size = sizes < 1000
>>> remove_pixel = mask_size[label_im]
>>> remove_pixel.shape
(256, 256)
>>> label_im[remove_pixel] = 0
>>> plt.imshow(label_im)
<matplotlib.image.AxesImage object at ...>
Ahora reasignamos etiquetas con np.searchsorted
>>> labels = np.unique(label_im)
>>> label_im = np.searchsorted(labels, label_im)
Encuentra una región de interes del objeto
>>> slice_x, slice_y = ndimage.find_objects(label_im==4)[0]
>>> roi = im[slice_x, slice_y]
>>> plt.imshow(roi)
<matplotlib.image.AxesImage object at ...>
Otras mediciones espaciales: ndimage.center_of_mass, ndimage.maximum_position, etc.
Pueden ser usadas fuera del limitado campo de acción de las aplicaciones de segmentación.
Ejemplo: bloque promedio
>>> from scipy import misc
>>> l = misc.lena()
>>> sx, sy = l.shape
>>> X, Y = np.ogrid[0:sx, 0:sy]
>>> regions = sy/6 * (X/4) + Y/6 # note that we use broadcasting
>>> block_mean = ndimage.mean(l, labels=regions, index=np.arange(1,
... regions.max() +1))
>>> block_mean.shape = (sx/4, sy/6)
Cuando las regiones son bloques regulares, es más eficiente usar stride tricks (Example: fake dimensions with strides).
Bloques no espaciados regularmente: promedio radial
>>> sx, sy = l.shape
>>> X, Y = np.ogrid[0:sx, 0:sy]
>>> r = np.hypot(X - sx/2, Y - sy/2)
>>> rbin = (20* r/r.max()).astype(np.int)
>>> radial_mean = ndimage.mean(l, labels=rbin, index=np.arange(1, rbin.max() +1))
Ejercicio: segmentación

Otras mediciones
Función de correlación, espectro de Fourier/wavelet, etc.
Un ejemplo con morfología matemática: granulometría (http://en.wikipedia.org/wiki/Granulometry_%28morphology%29)
>>> def disk_structure(n):
... struct = np.zeros((2 * n + 1, 2 * n + 1))
... x, y = np.indices((2 * n + 1, 2 * n + 1))
... mask = (x - n)**2 + (y - n)**2 <= n**2
... struct[mask] = 1
... return struct.astype(np.bool)
...
>>>
>>> def granulometry(data, sizes=None):
... s = max(data.shape)
... if sizes == None:
... sizes = range(1, s/2, 2)
... granulo = [ndimage.binary_opening(data, \
... structure=disk_structure(n)).sum() for n in sizes]
... return granulo
...
>>>
>>> np.random.seed(1)
>>> n = 10
>>> l = 256
>>> im = np.zeros((l, l))
>>> points = l*np.random.random((2, n**2))
>>> im[(points[0]).astype(np.int), (points[1]).astype(np.int)] = 1
>>> im = ndimage.gaussian_filter(im, sigma=l/(4.*n))
>>>
>>> mask = im > im.mean()
>>>
>>> granulo = granulometry(mask, sizes=np.arange(2, 19, 4))